Ich muss ja gestehen, dass ich ein wenig mit mir gehadert habe, welcher nächste Schritt in unserer Gentechnik-Serie der logische ist. Nachdem es ja im letzten Text grob um den Aufbau unserer DNA ging, hätte sich dieser Text mit der RNA beschäftigen können, dem kleinen Bruder der DNA. Oder ich schreibe darüber, was mit dem DNA-Strang eigentlich passiert, wenn er sich teilt. Meine Entscheidung fiel auf letzteres, also reden wir heute nochmal über DNA, wie sie sich teilt und vermehrt.
Und wir steigen auch direkt knallhart ein, deshalb lege ich euch nahe, zur Auffrischung nochmal den Text über Nucleinsäuren zu lesen. In diesem Text war zu lesen, dass jede Nukleinbase genau einen Partner hat, mit dem sie sich verbindet. Im Umkehrschluss bedeutet das, wenn ich eine Nukleinbase kenne, weiß ich auch ganz genau, welche andere Nukleinbase ich brauche, um ein Basenpaar zu formen. Und genau diesen Umstand macht man sich beim Aufteilen der DNA zunutze.
![I, Madprime [GFDL (https://www.gnu.org/copyleft/fdl.html), CC-BY-SA-3.0 or CC BY-SA 2.5-2.0-1.0], via Wikimedia Commons](https://nulliusinverba.blockblogs.de/wp-content/uploads/sites/3/2015/06/DNA-Replikation-Wozu-ist-DNA-eigentlich-gut-Bild-1-300x149.png)
Am Anfang kommt dafür ein Enzym ins Spiel. Was Enzyme sind, klären wir auch noch. Hier ist es erstmal wichtig zu wissen, dass ein Enzym namens „Helikase“ die Basenpaare praktisch zerschneidet. Man könnte auch sagen, dass es sie auftrennt oder aufschmilzt, aber ich stell‘ mir gerne vor, dass die Helikase – nichts anderes, als ein großes Molekül – vollkommen übergeschnappt und marodierend durch die Basenpaare wirbelt. Damit diese Helikase durch die Basenpaare, also die Nukleotide, hindurchschneiden kann, muss der DNA-Strang geradegezogen werden. Ungefähr so, als würde man lockige Haare mit einem Glätteisen (Word kennt diesen Begriff übrigens nicht. Ich bin erstaunt) bearbeiten. Die Funktion des Glätteisens übernimmt in der DNA ein Enzym namens „Topoisomerase“. Das ist allerdings jetzt eher nebensächlich, aber vielleicht kann der ein oder andere dieses Wissen ja gewinnbringend in sein/ihr Wochenende einbringen.
Nun betrachten wir eine weitere Grafik. Um euch nicht direkt zu sehr zu verwirren bitte ich, sämtliche Begriffe auf der Grafik zu ignorieren, die bisher im Text noch nicht besprochen wurden. Wir kommen noch zu allen.

Nun haben wir also einen ganzen DNA-Strang, der in zwei halbe aufgespalten wurde. Die beiden Nukleotidstränge, die aus dem ursprünglichen Strang aufgeteilt wurden, nennt man auch „Mutterstrang“. An diese beiden Mutterstränge wird jeweils ein komplementärer Nukleotidstrang angebracht. Diese angebrachten Stränge nennt man auch Tochterstrang. Nun kann man nicht einfach irgendwie einen Tochterstrang anfügen, wie man gerade lustig ist. Jetzt kommen die 3‘ und 5‘ (gesprochen: Drei Strich, sowie fünf Strich) Punkte ins Spiel. In meinem Text über Kokosöl haben wir damit begonnen, Kohlenstoffatome zu zählen. Und genau sowas machen wir jetzt auch wieder. Dieses Mal zählen wir allerdings die Kohlenstoffatome des Zuckermoleküls, das Teil jedes Nukleotids ist. Dieser Zucker sieht so aus:
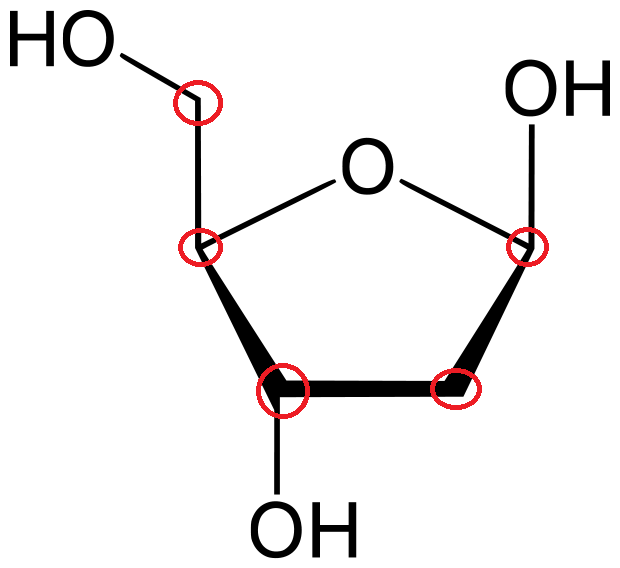
Die roten Kreise wurden von mir eingefügt und bezeichnen die Punkte, an denen sich die Kohlenstoffatome befinden. Und jetzt fangen wir im Uhrzeigersinn oben an, die Kohlenstoffatome zu zählen. Das 3‘-Atom befindet sich also links unten, das 5‘-Atom links oben. Das 3‘-Atom ist im Nukleotid der Anlaufpunkt für den Phosphor des vorherigen Nukleotids. Am 5‘-Atom befindet sich der Phosphor des jeweiligen Nukleotids. So bindet sich also immer der Phosphor vom 5‘-Ende an das 3‘-Ende des nächsten Nukleotids und so weiter und so fort.
Besonders schön zu sehen ist es in der folgenden Grafik, die auch schon in meinem letzten Text Verwendung fand:
![von DNA_chemical_structure.svg: Madeleine Price Ball, User:Madprime derivative work: Matt (Diskussion) (DNA_chemical_structure.svg) [CC BY-SA 2.5-2.0-1.0 (https://creativecommons.org/licenses/by-sa/2.5-2.0-1.0), GFDL (https://www.gnu.org/copyleft/fdl.html) oder CC-BY-SA-3.0 (https://creativecommons.org/licenses/by-sa/3.0/)], via Wikimedia Commons](https://nulliusinverba.blockblogs.de/wp-content/uploads/sites/3/2015/06/DNA-Replikation-Wozu-ist-DNA-eigentlich-gut-Bild-4.png)
Aber wie kommt denn jetzt eigentlich der Tochterstrang an den Mutterstrang?
Am Anfang steht der sogenannte Primer. Dieser besteht aus RNA-Nukleotiden. Was RNA ist, klären wir im nächsten Text, aber als kleine Einführung sei einfach gesagt, dass es zwischen DNA-Nukleotiden und RNA-Nukleotiden zwei größere Unterschiede gibt. Die Nukleotide der DNA sind eine der vier Basen (Adenin, Guanin, Cytosin, Thymin), zusammen mit dem Zuckermolekül und dem Phosphor. Die Nukleotide der RNA bestehen aus Adenin, Guanin, Cytosin und Uracil, sowie einem leicht anderen Zuckermolekül. Für den sogenannten Primer kommen bis zu 30 dieser Nukleotide zusammen.
Ein Enzym namens „Primase“ stellt die Primer her. Die Primase wird am 3‘-Ende in Richtung 5‘-Ende des Mutterstrangs befestigt und beginnt mit der Herstellung der Primer. Der Punkt, an dem der Primer sitzt, ist gleichzeitig der Startpunkt für die Herstellung des Tochterstrangs.
Jetzt kommt ein weiteres Enzym ins Spiel. Die sogenannte „DNA-Polymerase“. Dieses Enzym ist jetzt dafür verantwortlich, an jede Base im Mutterstrang, die entsprechende Base (sowie den Rest des Nukleotids) des Tochterstrangs anzubringen. Dabei stellt es die Basen allerdings nicht selbst her. Diese Basen befinden sich bereits im Cytoplasma und gelangen über sogenannte „Kernporen“ in den Zellkern. Die DNA-Polymerase stellt also den Tochterstrang her, und verbindet ihn mit dem Mutterstrang. Dabei arbeitet die Polymerase immer vom 3‘ zum 5‘ Ende hin.
An dieser Stelle sei schnell eine Kleinigkeit eingeworfen: Die Mutterstränge, die aus dem DNA-Strang entstehen, teilen sich nochmal in Leitstrang und Folgestrang auf. Der Leitstrang ist immer der mit dem 5‘-Ende auf der rechten Seite, der Folgestrang ist der mit dem 3‘-Ende auf der rechten Seite. Da die Polymerase immer vom 3‘-Ende zum 5‘-Ende läuft, bedeutet das, dass die Polymerase am Leitstrang von links nach rechts läuft und die Polymerase am Folgestrang von rechts nach links abläuft. Ich verweise mal wieder an die zweite Grafik.
Wichtig wird das aus folgendem Grund:
Die Polymerase des Leitstrangs läuft in Richtung der Helikase. Die Helikase wird sozusagen als Windschatten benutzt. Die Polymerase läuft auch genau so lange, wie die Helikase die Basenpaare zerteilt. Alles vollkommen problemlos, da sich Polymerase, Primer, Helikase alle von links nach rechts bewegen.
Der Folgestrang hat jetzt aber ein Problem. Bei dem bewegt sich die Polymerase ebenfalls vom 3‘- zum 5‘-Ende. Dummerweise steht die Polymerase beim Folgestrang leider auf dem Kopf, das heißt, sie bewegt sich von rechts nach links. Die Polymerase am Folgestrang müsste also anfangen, einen Nucleotidstrang zu bearbeiten, der noch gar nicht von der Helikase vorbereitet wurde.
Hier nochmal die zweite Grafik, damit ihr nicht immer hochscrollen müsst.

Die Polymerase unten (am Leitstrang) läuft von links nach rechts, die Polymerase oben (am Folgestrang) läuft von rechts nach links. Beim Leitstrang läuft’s problemlos, der Folgestrang bekommt ein paar kleine Probleme. Aber die Evolution hat auch dafür eine Lösung parat.
Am Folgestrang wird irgendwo mittig ein Primer gesetzt. Die Polymerase arbeitet sich von rechts nach links am Folgestrang entlang und sobald sie am Ende des Strangs angekommen ist, springt sie wieder zurück in Richtung Helikase, wo sich inzwischen ein weiterer Primer befindet. Von diesem aus arbeitet sich die Polymerase zum vorherigen Primer, springt Richtung Helikase zum nächsten Primer und vollzieht dieses Spiel bis zum Ende des Stranges.
Betrachten wir jetzt mal auf der Grafik die sogenannten „Okazaki-Fragmente“. Diese sind die Strecke zwischen zwei Primern, die von der Polymerase abgearbeitet werden. Betrachtet man die Grafik genau, fällt auf, dass zwischen diesen Okazaki-Fragmenten kleine Unterbrechungen zu sehen sind. Das ist eine der Schwachstellen, die bei dieser Art von DNA-Reproduktion. Zwischen dem Anfang des ersten Okazaki-Fragments und dem Ende des nächsten, kann es passieren, dass eine oder mehrere Nukleotide fehlen. Außerdem sind die Primer ebenfalls noch in den Tochtersträngen vorhanden. Jetzt muss also jemand die Reste der Primer wegräumen und irgendwer sollte außerdem die DNA-Stränge reparieren.
Das Enzym RNase H schmeißt die Primer von den Strängen, eine weitere Polymerase (die DNA-Polymerase Alpha) rutscht über den Strang drüber und füllt die verbliebenen Lücken der Basenpaare.
Und zu guter Letzt ist ein weiteres Enzym an der Reihe. Die Sogenannte DNA-Ligase verknüpft nun noch die Lücken zwischen den Okazaki-Fragmenten, die entstanden sind, als die Primer entfernt wurden (denn man will ja anstelle der Primer wieder den Zucker und den Phosphor als „Rückgrat“ der DNA haben).
Nun wurden also mithilfe von jeder Menge Enzyme aus einem DNA-Strang zwei. Und damit wären wir schon am Ende des Textes angekommen, auch wenn es noch viel mehr zu dem Thema zu sagen gäbe. Wie man z.B. Fehler in diesem Replikationsvorgang vermeidet, ist aber Stoff für einen anderen Text.
Ich hoffe, der Artikel war verständlich. Er bildet nämlich praktisch die Grundlage für die Editierung von Genen und ist somit extrem wichtig, wenn es darum geht, Gentechnik verstehen zu wollen. Sollte es also bei der Verständlichkeit irgendwo Probleme geben, scheut euch nicht, einen Kommentar zu hinterlassen, ich helfe liebend gerne dabei, alle Unklarheiten zu beseitigen J
*Eigentlich wollte ich den Text ja „Two roads diverged in a yellow wood“ betiteln. Aber ich war mir nicht sicher, wie viele Leute die Referenz verstanden hätten.
Zeile 7: „In diesem Text war zu lesen, dass jede Aminosäure genau einen Partner hat, mit dem sie sich verbindet. Im Umkehrschluss bedeutet das, wenn ich eine Aminosäure kenne, weiß ich auch ganz genau, welche andere Aminosäure ich brauche, um ein Basenpaar zu formen.“
Aminosäure??? Gemeint ist wohl Nukleinbase.
Selbstverständlich. Wie kam ich denn da auf Aminosäure?
Das kommt wohl davon, wenn man mitten in der Nacht noch Texte schreibt…